Biología Molecular
Definición
Biología molecular / m ə l ɛ k j ʊ l ər / es una rama de la biología que se refiere a la base molecular de la actividad biológica entre biomoléculas en los diversos sistemas de una célula, incluyendo las interacciones entre ADN, ARN, proteínas y su biosíntesis, así como la regulación de estas interacciones. Escribiendo en Nature en 1961, William Astbury describió la biología molecular como:Relación con otras ciencias biológicas
Los investigadores en biología molecular utilizan técnicas específicas nativas de la biología molecular, pero las combinan cada vez más con técnicas e ideas de la genética y la bioquímica. No hay una línea definida entre estas disciplinas. La figura de la derecha es un esquema que representa una posible vista de las relaciones entre los campos:
- La bioquímica es el estudio de las sustancias químicas y los procesos vitales que ocurren en los organismos vivos. Los bioquímicos se centran principalmente en el papel, la función y la estructura de las biomoléculas. El estudio de la química detrás de los procesos biológicos y la síntesis de moléculas biológicamente activas son ejemplos de bioquímica.
- La genética es el estudio del efecto de las diferencias genéticas en los organismos. Esto a menudo puede inferirse por la ausencia de un componente normal (por ejemplo, un gen). El estudio de "mutantes" - organismos que carecen de uno o más componentes funcionales con respecto al llamado "tipo salvaje" o fenotipo normal. Las interacciones genéticas (epistasis) a menudo pueden confundir interpretaciones simples de tales estudios "knockout".
- La biología molecular es el estudio de las bases moleculares de los procesos de replicación, transcripción, traducción y función celular. El dogma central de la biología molecular, en donde el material genético se transcribe en ARN y luego se traduce en proteína, a pesar de ser simplificado en exceso, todavía proporciona un buen punto de partida para comprender el campo. La imagen ha sido revisada a la luz de las nuevas funciones emergentes para el ARN.
Gran parte de la biología molecular es cuantitativa, y recientemente se ha trabajado mucho en su interfaz con la informática en bioinformática y biología computacional. A principios de la década de 2000, el estudio de la estructura y función de los genes, la genética molecular, ha estado entre los subcampos más destacados de la biología molecular. Cada vez más áreas de biología se enfocan en moléculas, ya sea estudiando directamente las interacciones en biología celular y biología del desarrollo, o indirectamente, donde las técnicas moleculares se utilizan para inferir atributos históricos de poblaciones o especies, como en los campos de la biología evolutiva como la genética de poblaciones y la filogenética. También existe una larga tradición de estudiar biomoléculas "desde cero" en biofísica.
Técnicas de biología molecular
Clonación molecular
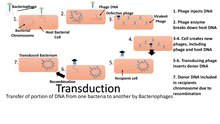
Una de las técnicas más básicas de biología molecular para estudiar la función proteica es la clonación molecular. En esta técnica, el ADN que codifica una proteína de interés se clona usando la reacción en cadena de la polimerasa (PCR) y / o enzimas de restricción en un plásmido (vector de expresión). Un vector tiene 3 características distintivas: un origen de replicación, un sitio de clonación múltiple (MCS) y un marcador selectivo, generalmente resistencia a antibióticos. Localizados corriente arriba del sitio de clonación múltiple están las regiones promotoras y el sitio de inicio de la transcripción que regulan la expresión del gen clonado. Este plásmido se puede insertar en células bacterianas o animales. La introducción de ADN en las células bacterianas puede realizarse por transformación a través de la captación de ADN desnudo, la conjugación a través del contacto célula-célula o mediante transducción a través de un vector viral. Introduciendo ADN en células eucariotas, como células animales, por medios físicos o químicos se llama transfección. Están disponibles varias técnicas de transfección diferentes, tales como la transfección de fosfato de calcio, la electroporación, la microinyección y la transfección de liposomas. El plásmido puede integrarse en el genoma, dando como resultado una transfección estable, o puede permanecer independiente del genoma, denominado transfección transitoria.
El ADN que codifica una proteína de interés se encuentra ahora dentro de una célula, y la proteína ahora se puede expresar. Una variedad de sistemas, tales como promotores inducibles y factores específicos de señalización celular, están disponibles para ayudar a expresar la proteína de interés a altos niveles. Se pueden extraer grandes cantidades de una proteína de la célula bacteriana o eucariota. La proteína puede analizarse para determinar la actividad enzimática en una variedad de situaciones, la proteína puede cristalizarse para estudiar su estructura terciaria o, en la industria farmacéutica, estudiar la actividad de nuevos fármacos contra la proteína.
Reacción en cadena de la polimerasa
La reacción en cadena de la polimerasa (PCR) es una técnica extremadamente versátil para copiar ADN. En resumen, la PCR permite que una secuencia de ADN específica se copie o modifique de formas predeterminadas. La reacción es extremadamente poderosa y en condiciones perfectas podría amplificar una molécula de ADN para convertirse en 1.07 billones de moléculas en menos de dos horas. La técnica de PCR puede usarse para introducir sitios de enzimas de restricción en los extremos de moléculas de ADN, o para mutar bases particulares de ADN, este último es un método denominado mutagénesis dirigida al sitio. La PCR también puede usarse para determinar si se encuentra un fragmento de ADN particular en una biblioteca de ADNc. La PCR tiene muchas variaciones, como la PCR de transcripción inversa (RT-PCR) para la amplificación de ARN y, más recientemente, la PCR cuantitativa que permite la medición cuantitativa de moléculas de ADN o ARN.
Electroforesis en gel
La electroforesis en gel es una de las herramientas principales de la biología molecular. El principio básico es que el ADN, el ARN y las proteínas se pueden separar por medio de un campo y tamaño eléctricos. En la electroforesis en gel de agarosa, el ADN y el ARN se pueden separar en función del tamaño haciendo pasar el ADN a través de un gel de agarosa con carga eléctrica. Las proteínas se pueden separar en función del tamaño mediante el uso de un gel SDS-PAGE, o en función del tamaño y su carga eléctrica mediante el uso de lo que se conoce como electroforesis en gel 2D.
Macromolécula secante y sondeo
Los términos Northern , Western y Eastern Blotting se derivan de lo que inicialmente era una broma de biología molecular que jugó con el término Southern blot , después de la técnica descrita por Edwin Southern para la hibridación de ADN borrado. Patricia Thomas, desarrolladora de la transferencia de ARN que luego se conoció como la mancha del norte , en realidad no usó el término.
Southern blot
Lleva el nombre de su inventor, el biólogo Edwin Southern, el Southern blot es un método para investigar la presencia de una secuencia específica de ADN dentro de una muestra de ADN. Las muestras de ADN antes o después de la digestión con la enzima de restricción (endonucleasa de restricción) se separan mediante electroforesis en gel y luego se transfieren a una membrana mediante transferencia por acción capilar. La membrana luego se expone a una sonda de ADN marcada que tiene una secuencia de base de complemento a la secuencia en el ADN de interés. La transferencia de Southern se utiliza con menos frecuencia en la ciencia de laboratorio debido a la capacidad de otras técnicas, como la PCR, para detectar secuencias de ADN específicas de muestras de ADN. Sin embargo, estas transferencias todavía se usan para algunas aplicaciones, tales como la medición del número de copias transgénicas en ratones transgénicos o en la ingeniería de líneas de células madre embrionarias genéticamente inactivadas.
Northern blot
La transferencia Northern se usa para estudiar los patrones de expresión de un tipo específico de molécula de ARN como comparación relativa entre un conjunto de diferentes muestras de ARN. Es esencialmente una combinación de electroforesis en gel de RNA desnaturalizante y una transferencia. En este proceso, el ARN se separa en función del tamaño y luego se transfiere a una membrana que luego se prueba con un complemento marcado de una secuencia de interés. Los resultados se pueden visualizar a través de una variedad de formas dependiendo de la etiqueta utilizada; sin embargo, la mayoría resulta en la revelación de bandas que representan los tamaños del ARN detectado en la muestra. La intensidad de estas bandas está relacionada con la cantidad de ARN objetivo en las muestras analizadas. El procedimiento se usa comúnmente para estudiar cuándo y cuánta expresión génica se produce al medir qué tanto de ese ARN está presente en diferentes muestras.
Western Blot
En el western blot, las proteínas se separan primero por tamaño, en un gel delgado intercalado entre dos placas de vidrio en una técnica conocida como SDS-PAGE. Las proteínas en el gel se transfieren a un fluoruro de polivinilideno (PVDF), nitrocelulosa, nylon u otra membrana de soporte. Esta membrana puede ser probada con soluciones de anticuerpos. Los anticuerpos que se unen específicamente a la proteína de interés se pueden visualizar mediante una variedad de técnicas, que incluyen productos coloreados, quimioluminiscencia o autorradiografía. A menudo, los anticuerpos están etiquetados con enzimas. Cuando un sustrato quimioluminiscente se expone a la enzima, permite la detección. El uso de técnicas de western blot permite no solo la detección sino también el análisis cuantitativo. Se pueden usar métodos análogos a la transferencia de Western para teñir directamente proteínas específicas en células vivas o secciones de tejido.
Transferencia del este
La técnica de transferencia del este se usa para detectar la modificación postraduccional de las proteínas. Las proteínas transferidas a la membrana de PVDF o de nitrocelulosa son probadas para modificaciones usando sustratos específicos.
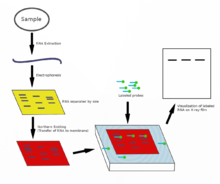
Microarrays
El microarreglo de ADN es una colección de manchas unidas a un soporte sólido tal como un portaobjetos de microscopio donde cada punto contiene uno o más fragmentos de oligonucleótidos de ADN monocatenarios. Las matrices permiten colocar grandes cantidades de puntos muy pequeños (100 micras de diámetro) en una sola diapositiva. Cada punto tiene una molécula de fragmento de ADN que es complementaria a una única secuencia de ADN. Una variación de esta técnica permite calificar la expresión génica de un organismo en una etapa particular del desarrollo (perfil de expresión). En esta técnica, el ARN en un tejido se aísla y se convierte en ADNc marcado. Este ADNc se hibrida luego con los fragmentos en la matriz y se puede realizar la visualización de la hibridación. Como se pueden hacer múltiples matrices con exactamente la misma posición de fragmentos, son particularmente útiles para comparar la expresión génica de dos tejidos diferentes, como un tejido sano y canceroso. Además, uno puede medir qué genes se expresan y cómo esa expresión cambia con el tiempo o con otros factores. Hay muchas formas diferentes de fabricar microarrays; los más comunes son chips de silicio, portaobjetos de microscopio con manchas de ~ 100 micras de diámetro, arreglos personalizados y arreglos con manchas más grandes en las membranas porosas (macroarrays). Puede haber desde 100 puntos hasta más de 10,000 en un conjunto determinado. Las matrices también se pueden hacer con moléculas distintas de ADN. Hay muchas formas diferentes de fabricar microarrays; los más comunes son chips de silicio, portaobjetos de microscopio con manchas de ~ 100 micras de diámetro, arreglos personalizados y arreglos con manchas más grandes en las membranas porosas (macroarrays). Puede haber desde 100 puntos hasta más de 10,000 en un conjunto determinado. Las matrices también se pueden hacer con moléculas distintas de ADN. Hay muchas formas diferentes de fabricar microarrays; los más comunes son chips de silicio, portaobjetos de microscopio con manchas de ~ 100 micras de diámetro, arreglos personalizados y arreglos con manchas más grandes en las membranas porosas (macroarrays). Puede haber desde 100 puntos hasta más de 10,000 en un conjunto determinado. Las matrices también se pueden hacer con moléculas distintas de ADN.
Oligonucleótido alelo-específico
El oligonucleótido aleloespecífico (ASO) es una técnica que permite la detección de mutaciones de base única sin la necesidad de PCR o electroforesis en gel. Las sondas marcadas cortas (20-25 nucleótidos de longitud) se exponen al ADN objetivo no fragmentado, la hibridación ocurre con alta especificidad debido a la corta longitud de las sondas e incluso un solo cambio de base impedirá la hibridación. El ADN objetivo se lava luego y las sondas marcadas que no se hibridaron se eliminan. El ADN diana se analiza a continuación para la presencia de la sonda a través de radioactividad o fluorescencia. En este experimento, como en la mayoría de las técnicas de biología molecular, se debe usar un control para asegurar una experimentación exitosa.
En biología molecular, los procedimientos y las tecnologías se desarrollan continuamente y las tecnologías más antiguas se abandonan. Por ejemplo, antes del advenimiento de la electroforesis en gel de ADN (agarosa o poliacrilamida), el tamaño de las moléculas de ADN se determinaba típicamente por velocidad de sedimentación en gradientes de sacarosa, una técnica lenta y laboriosa que requiere una costosa instrumentación; antes de gradientes de sacarosa, se usó viscometría. Además de su interés histórico, a menudo vale la pena conocer la tecnología más antigua, ya que ocasionalmente es útil resolver otro nuevo problema para el cual la técnica más nueva es inapropiada.
Historia
Mientras que la biología molecular se estableció en la década de 1930, Warren Weaver acuñó el término en 1938. Weaver era el director de Ciencias Naturales de la Fundación Rockefeller en ese momento y creía que la biología estaba a punto de sufrir un cambio significativo dados los recientes avances en campos como la cristalografía de rayos X.
La investigación clínica y las terapias médicas derivadas de la biología molecular están parcialmente cubiertas por la terapia genética. El uso de enfoques de biología molecular o biología celular molecular en medicina ahora se llama medicina molecular. La biología molecular también juega un papel importante en la comprensión de las formaciones, acciones y regulaciones de varias partes de las células que pueden usarse para apuntar de manera eficiente a nuevas drogas, diagnosticar enfermedades y comprender la fisiología de la célula.